Charme: la molecola di lncRNA che controlla lo sviluppo del cuore
Charme è un lncRNA che controlla lo sviluppo cardiaco attraverso circuiti molecolari che si instaurano nel muscolo grazie alla sua interazione con la proteina Matrin3
La molecola di RNA è in grado di costruire specifiche reti di interazione per un controllo temporale e spaziale dei processi di formazione del cuore.
È quanto dimostrato da un nuovo studio coordinato dal Dipartimento di Biologia e biotecnologie “Charles Darwin” della Sapienza e pubblicato sulla rivista eLife.

Per affrontare la complessità dei processi biologici, le cellule sfruttano molteplici sistemi di regolazione, spesso basati sull’attività di molecole di RNA, come nel caso dei lunghi RNA non codificanti (long non coding RNA, lncRNA) che non producono proteine. Queste molecole sono in grado di costruire specifiche reti di interazione per un controllo temporale e spaziale dei processi biologici.
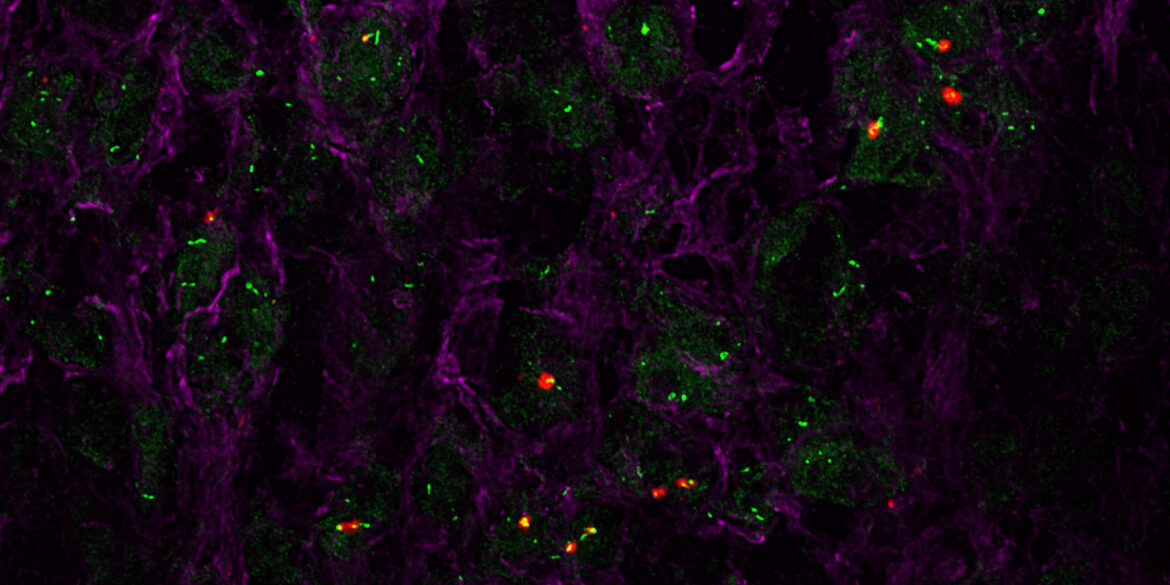
È il caso di Charme, un lncRNA che controlla lo sviluppo cardiaco attraverso circuiti molecolari che si instaurano nel muscolo grazie alla sua interazione con la proteina Matrin3. Matrin3 è coinvolta in diverse miopatie e in malattie neurodegenerative, come la Sclerosi laterale amiotrofica (SLA).
Un nuovo studio italiano pubblicato sulla rivista internazionale eLife e coordinato dal Dipartimento di Biologia e biotecnologie “Charles Darwin” della Sapienza in collaborazione con l’Istituto italiano di tecnologia e l’European Molecular Biology Laboratory ha rivelato il ruolo chiave di Charme nell’accensione di geni necessari alla maturazione delle cellule del cuore. La presenza di Charme già durante le fasi embrionali dello sviluppo cardiaco, si è rivelata fondamentale per guidare Matrin3 sui giusti contesti genomici, promuovendo la funzionalità e lo sviluppo cardiaco.
“Tra i piani futuri del laboratorio – spiega Monica Ballarino della Sapienza – c’è l’ulteriore caratterizzazione funzionale di Charme che è abbondantemente espresso nel muscolo umano. Questo permetterà una migliore comprensione della fisiologia e dello sviluppo del cuore ed il disegno di nuove strategie diagnostiche e terapeutiche per le patologie cardiache.”
Riferimenti:
The long noncoding RNA Charme supervises cardiomyocyte maturation by controlling cell differentiation programs in the developing heart – Valeria Taliani, Giulia Buonaiuto, Fabio Desideri, Adriano Setti, Tiziana Santini, Silvia Galfrè, Leonardo Schirone, Davide Mariani, Giacomo Frati, Valentina Valenti, Sebastiano Sciarretta, Emerald Perlas, Carmine Nicoletti, Antonio Musarò, Monica Ballarino – eLife 2023 https://doi.org/10.7554/eLife.81360
Testo dal Settore Ufficio stampa e comunicazione Sapienza Università di Roma