Nanostelle per una nuova tecnologia che imita l’organizzazione cellulare: DNA e anticorpi insieme per una svolta nei biomateriali intelligenti
Roma, 4 novembre 2025 – Una collaborazione tra ricercatori delle Università di Roma Tor Vergata e Sapienza ha portato allo sviluppo di una nuova e promettente tecnologia che combina la nanotecnologia del DNA con la precisione degli anticorpi, le molecole naturali che riconoscono specifici bersagli. Lo studio è pubblicato sul Journal of the American Chemical Society (JACS).

Un team di ricercatori dell’Università degli Studi di Roma Tor Vergata, in collaborazione con Sapienza Università di Roma, ha sviluppato un nuovo sistema molecolare in grado di creare micro-compartimenti programmabili che imitano il modo in cui le cellule organizzano le proprie funzioni interne. Lo studio è stato pubblicato su JACS – Journal of the American Chemical Society, una tra le riviste più autorevoli nel campo della chimica.
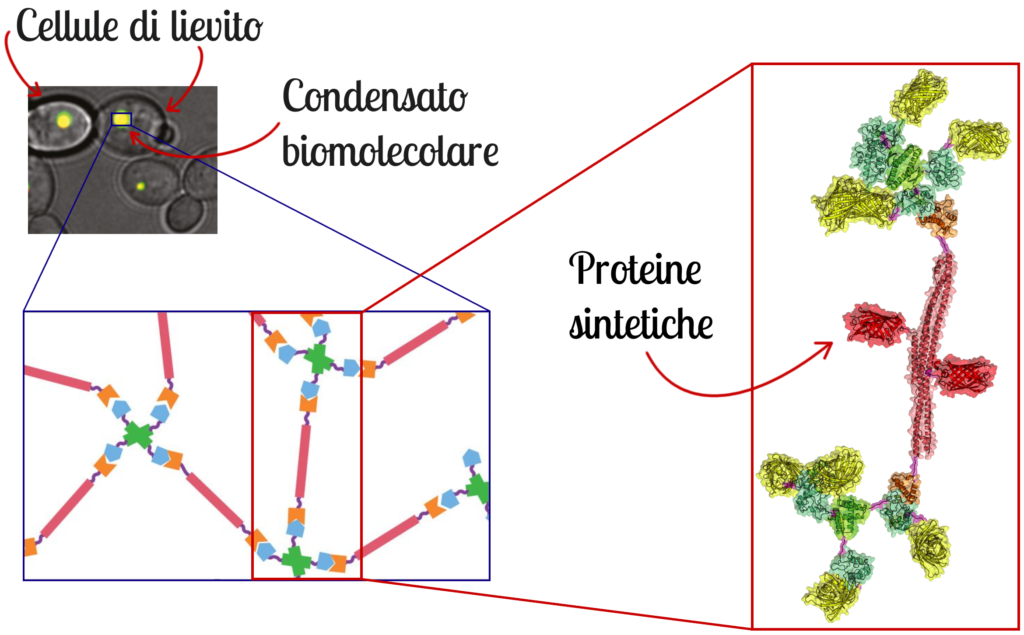
Alla base di questa tecnologia ci sono strutture chiamate nanostelle di DNA, molecole di DNA sintetico progettate in laboratorio con quattro bracci. Tre terminano con sequenze adesive che permettono alle nanostelle di riconoscersi e agganciarsi tra loro in modo controllato. Il quarto braccio è invece modificato con un antigene, cioè una porzione molecolare riconosciuta in modo specifico da un anticorpo.
Quando è presente l’anticorpo corretto, questo si lega agli antigeni di nanostelle diverse e funziona come un ponte molecolare, collegandole tra loro e facendo nascere i micro-compartimenti sferici. È proprio la presenza dell’anticorpo a “decidere” quando questi compartimenti si formano, si dissolvono o si riformano: per questo il sistema è programmabile. Variando tipo e quantità di anticorpo, i ricercatori possono controllare in modo preciso il comportamento del sistema.
La parte innovativa di questo lavoro consiste nel dimostrare che DNA e anticorpi possono essere usati insieme come elementi costruttivi per creare micro-strutture dinamiche che riproducono artificialmente una logica tipica dei sistemi biologici: formare ambienti interni altamente regolati in risposta a un segnale specifico. Questo meccanismo è alla base dell’organizzazione cellulare naturale e rappresenta uno dei tratti più complessi da imitare in laboratorio.
“Questa scoperta apre porte a possibilità entusiasmanti. La capacità dei micro-compartimenti di formarsi in risposta a molecole specifiche, per esempio, potrebbe essere utilizzata per rilevare marcatori biologici, permettendo nuovi strumenti diagnostici,” afferma Erica Del Grosso, ricercatrice principale del progetto e professoressa associata presso il Dipartimento di Scienze e tecnologie chimiche dell’Università di Roma Tor Vergata”.
“I nostri micro-compartimenti ibridi anticorpo-DNA sono come un ponte tra biologia e tecnologia,” conferma Francesco Ricci, ricercatore principale del progetto e professore ordinario presso lo stesso Dipartimento, “Non sono solo stabili e precisi, ma programmabili, fornendo un modo per creare strutture artificiali simili a cellule e sostenere la ricerca di nuovi biomateriali.”
“Unire DNA e anticorpi è come costruire un ponte tra due mondi: scoprire come queste molecole interagiscono e collaborano apre nuove prospettive e rende la ricerca che faccio sempre più stimolante”. Aggiunge Sara Scalia, primo autore dell’articolo e dottoranda presso il gruppo guidato dal Prof. Francesco Ricci.
“Dal punto di vista teorico siamo riusciti a sviluppare un modello che ha permesso di spiegare l’origine dei micro-compartimenti e di prevederne il comportamento” conclude Lorenzo Rovigatti, professore di fisica della materia teorica presso la Sapienza Università di Roma, “un passo importante verso lo sviluppo di applicazioni in ambito biomedico e della scienza dei materiali.”
Il progetto è stato sostenuto dal Consiglio europeo per la ricerca (ERC), dall’Associazione italiana per la ricerca sul cancro (AIRC), dal Ministero dell’Università e della ricerca (MUR) e da NextGenerationEU – Missione 4, Componente 2.
Riferimenti bibliografici:
Sara Scalia, Marco Cappa, Lorenzo Rovigatti, Erica Del Grosso e Francesco Ricci, Immune-Induced Antibody–DNA Hybrid Condensates, Journal of the American Chemical Society, Articles ASAP (Article) –
October 23, 2025 – DOI: 10.1021/jacs.5c13855
Testo dal Settore Ufficio stampa e comunicazione Sapienza Università di Roma e dall’Ufficio Stampa dell’Università degli Studi di Roma Tor Vergata