Combattere le metastasi del tumore al colon-retto: dimostrato il ruolo dell’alleanza tra macrofagi e le cellule “natural killer”
In uno studio condotto alla Sapienza Università di Roma, un gruppo di ricercatori ha dimostrato il ruolo chiave dell’alleanza tra macrofagi e cellule “natural killer” per contrastare la crescita delle metastasi. I risultati, pubblicati sul “Journal of Clinical Investigation” aprono la strada a nuove terapie per il tumore al colon-retto
Il cancro del colon-retto (CRC) è la seconda causa di morte per tumore al mondo. Nonostante sia divenuto molto più curabile negli ultimi anni, le terapie non sono ancora efficaci in tutti i pazienti. L’ancora elevato tasso di mortalità per questo tipo di tumore è dovuto soprattutto alla capacità di diffondersi in altre parti del corpo, facendo insorgere metastasi in una rilevante frazione di pazienti.
In questo contesto, le cellule “natural killer” (NK) sono un tipo di globuli bianchi appartenenti al sistema immunitario innato, che possono aiutare a contrastare la diffusione del tumore. Il loro nome deriva proprio dalla capacità di riconoscere e distruggere spontaneamente le cellule tumorali e inibire la formazione di metastasi. Nonostante ciò, rimane ancora poco chiaro il comportamento delle cellule “natural killer” a metastasi già formate, in particolare nel fegato.
In una ricerca sostenuta dalla Fondazione AIRC per la ricerca sul cancro, l’immunologo Giovanni Bernardini e colleghi hanno studiato il ruolo specifico delle cellule NK nell’ambiente metastatico, per comprendere come proteggerne e preservarne meglio le capacità anti-tumorali. Coordinata dal Dipartimento di Medicina Molecolare della “Sapienza”, la ricerca è avvenuta in collaborazione con diversi gruppi nazionali e internazionali. I risultati, pubblicati sul Journal of Clinical Investigation, hanno mostrato un meccanismo naturale di difesa che può aiutare a rallentare la crescita delle metastasi epatiche.
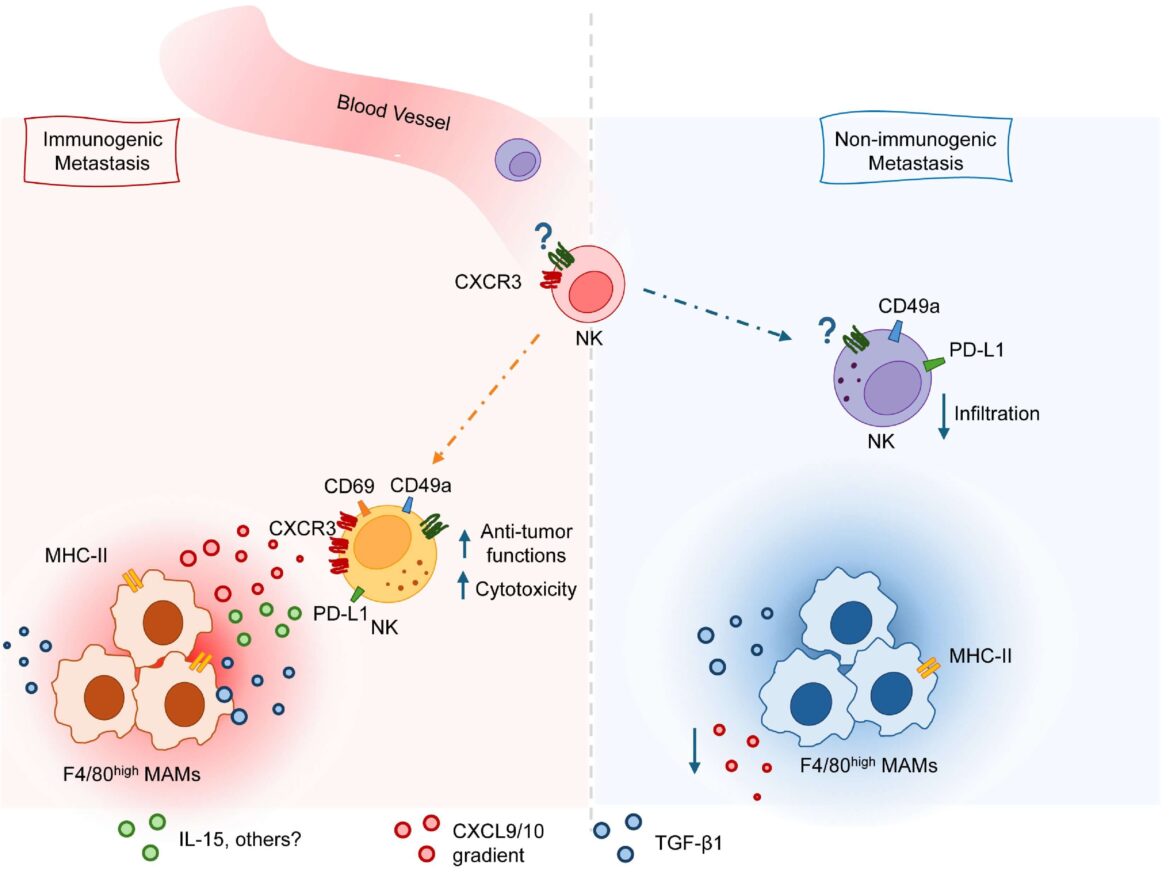
In particolare il gruppo guidato da Bernardini, professore di Immunologia presso “Sapienza”, ha dimostrato che non tutte le metastasi al fegato sono uguali. Alcuni tipi formano dei microambienti in cui l’attività delle cellule NK è potenziata, grazie alla presenza di molecole in grado di promuovere la persistenza di queste cellule e la loro capacità di attaccare il tumore.
Nello studio i ricercatori hanno analizzato metastasi epatiche ottenute sia da topi sia da biopsie di pazienti, trovando in entrambi i casi che un tipo specifico di macrofagi, i cosiddetti “spazzini” del sistema immunitario, è in grado di insegnare alle cellule “natural killer” come attivarsi correttamente e attaccare le cellule tumorali.
“Per fare ciò – spiega Giovanni Bernardini – i macrofagi producono due chemochine. Si tratta di particolari proteine, chiamate CXCL9 e CXCL10, che, come dei messaggeri chimici, attirano le cellule NK all’interno delle metastasi e creano un ambiente che permette a queste cellule di attivarsi”.
Alcuni risultati raccolti evidenziano la notevole importanza delle chemochine CXCL9 e CXCL10. In particolare, in topi con un deficit del recettore CXCR3, che permette di “sentire” gli effetti di queste due proteine, si osserva una inibizione delle cellule “natural killer” e la successiva accelerazione della crescita delle metastasi.

Nel complesso i dati pubblicati dimostrano che una corretta cooperazione tra macrofagi e cellule “natural killer” è fondamentale per attivare una risposta immunitaria in grado di limitare la diffusione del tumore del colon-retto, aprendo nuove prospettive terapeutiche basate sul potenziamento di questo network.
Riferimenti bibliografici:
Russo, C. D’Aquino, C. Di Censo, M. Laffranchi, L. Tomaipitinca, V. Licursi, S. Garofalo, J. Promeuschel, G. Peruzzi, F. Sozio, A. Kaffke, C. Garlanda, U. Panzer, C. Limatola, C.A.J. Vosshenrich, S. Sozzani, G. Sciumè, A. Santoni, G. Bernardini, Cxcr3 promotes protection from colorectal cancer liver metastasis by driving NK cell infiltration and plasticity, Journal of Clinical Investigation, 2025;135(11):e184036, DOI: https://doi.org/10.1172/JCI184036
Testo e immagine dal Settore Ufficio stampa e comunicazione Sapienza Università di Roma